
DEPARTAMENTO DE BIOQUÍMICA E BIOLOGIA MOLECULAR – ICB/UFG
PROF. CARLOS EDUARDO. - Curso: FARMÁCIA
SODRÉ NETO
EXERCÍCIO SOBRE TRANSCRIÇÃO E TRADUÇÃO:
Veja a escala de tamanho dos componentes aqui estudados
Parte 1 – replicação do DNA
1. Na sua opinião, qual experimento definiu que o DNA era o material genético? Justifique sua resposta.
Resposta : O experimento de Alvery onde se percebeu que quando se degradava DNA o efeito transformante se anulava , o que não ocorria quando se degradava proteínas e RNA.
Griffith descobriu que havia um principio transformante , Alvery e equipe definiram que era o DNA com o experimento de Alvery, e Hershey e Chase confirmaram e retiraram as dúvidas.
Experiências conduzidas pelo microbiólogo inglês Frederick Griffith haviam demonstrado que quando um extracto morto da estirpe lisa era misturado com a estirpe rugosa viva e injectada num rato, os tecidos do animal passavam a conter uma estirpe viva e lisa. A maioria dos cientistas teorizava que a mudança deveria ser provocada por uma proteína mas, após repetir a experiência muitas vezes, entre 1932 e 1944, Avery provou que era o Ácido Desoxirribonucleico (DNA) o responsável pela transferência de material genético entre células num processo chamado "transformação". A descoberta sugeria que o DNA seria o material genético básico da célula, facto que veio a ser confirmado por cientistas posteriores. O trabalho de Avery inspirou várias pesquisas sobre a estrutura do DNA, agora conhecida como código genético.
Experimento de Oswald Avery in vitro, através do qual ele conseguiu identificar o princípio transformador ou a substância química que promove a transformação
 Leia mais: https://geneticavirtual.webnode.com.br/genetica-virtual-home/prefacio/estrutura%20e%20replica%c3%a7%c3%a3o%20do%20dna/identifica%c3%a7%c3%a3o%20do%20principio%20transformante/
Leia mais: https://geneticavirtual.webnode.com.br/genetica-virtual-home/prefacio/estrutura%20e%20replica%c3%a7%c3%a3o%20do%20dna/identifica%c3%a7%c3%a3o%20do%20principio%20transformante/
2. Quais diferenças que podemos relacionar entre o DNA e o RNA?

A principal diferença entre esses dois ácidos nucleicos é que o DNA é o responsável pelo armazenamento da informação genética utilizada no desenvolvimento dos organismos vivos, enquanto o RNA é o responsável por sintetizar proteínas.
Em sua estrutura, eles se diferem pois o DNA possui duas cadeias helicoidais, enquanto o RNA possui apenas uma cadeia. Porém, o RNA é mais versátil do que o DNA, sendo capaz de realizar inúmeras tarefas em um organismo.
| |
DNA |
RNA |
| Definição |
O DNA, ou ADN em português, é um composto orgânico cujas moléculas contêm as instruções genéticas que coordenam o desenvolvimento e funcionamento de todos os seres vivos e de alguns vírus.
Os genes do DNA são expressos através das proteínas que seus nucleotídeos produzem com a ajuda do RNA
|
RNA é um ácido nucleico responsável pela síntese de proteínas da célula.
As várias formas de RNA sintetizam essas proteínas de acordo com as informações contidas no DNA
|
| Significado |
Ácido desoxirribonucleico |
Ácido ribonucleico |
| Função |
Armazenar informação genética, controlar atividade celular e produzir RNA |
Sintetizar proteínas e transferir informação do DNA até o local de síntese de proteínas na célula |
| Estrutura |
Duas cadeias helicoidais. Suas cadeias são longas e possuem milhares de nucleótidos |
Uma cadeia. Possui cadeia curta com centenas de nucleótidos |
| Tipo de açúcar (pentose) |
Açúcar desoxirribose |
Açúcar ribose |
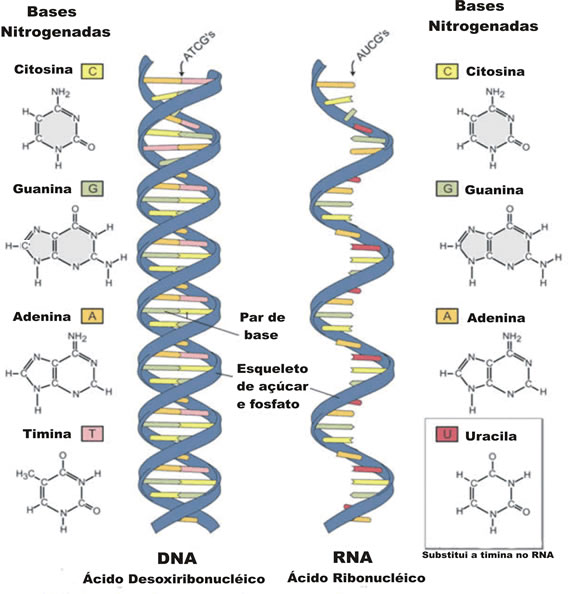
| Bases nitrogenadas |
As bases são citosina, guanina, adenina e timina |
As bases são citosina, guanina, adenina e uracila |
| Estabilidade química |
Muito estável |
Pouco estável |
| Origem |
Replicação |
Transcrição |
| Resistência |
Mais resistente à hidrólise |
Menos resistente à hidrólise |
| Forma |
Apresenta uma forma básica |
Pode ser mensageiro, transportador ou ribossômico |
| Localização |
O DNA é encontrado no núcleo de uma célula. Existe também o DNA mitocondrial, que se encontra na mitocôndria |
Varia de acordo com o tipo de RNA, podendo ser encontrado no núcleo de uma célula, no citoplasma e no ribossomo |
| Replicação |
O DNA é auto-replicante |
O RNA é sintetizado a partir do DNA |
Estruturas do RNA e DNA

DNA e RNA são ácidos nucleicos encontrados em praticamente todas as células humanas. Eles são responsáveis pela transmissão de caracteres hereditários e pela produção de proteínas compostas, que são o principal constituinte dos seres vivos.
DNA, que significa Ácido desoxirribonucleico, são as moléculas que contêm as instruções genéticas que coordenam o desenvolvimento e funcionamento de todos os seres vivos e de alguns vírus. Ele é encontrado no núcleo de uma célula (DNA nuclear) ou nas mitocôndrias (DNA mitocondrial).
O DNA é formado por duas cadeias na forma de uma dupla hélice, que são constituídas por um açúcar, um grupo fosfato e pelas bases nitrogenadas, que podem ser a Citosina, Guanina, Adenina e Timina.
A dupla hélice é um fator essencial na replicação do DNA durante a divisão celular, onde cada hélice faz uma cópia de si mesmo.
Durante a transcrição do DNA, o RNA é formado. Essa molécula é complementar ao DNA, ajudando a realizar as funções do DNA.
Ele possui uma cadeia simples, constituída por uma ribose (açúcar), por um fosfato e bases nitrogenadas, que podem ser a Citosina, Guanina, Adenina e Uracila.
Em ambos, as bases nitrogenadas estão ligadas ao esqueleto de açúcar e fosfato. Porém, no DNA, cada base é atribuída à uma base nitrogenada parceira na segunda fita. A adenina se liga à timina e a citosina à guanina.
No RNA, a adenina se liga à uracila, enquanto a citosina se liga à guanina. Como ela é uma molécula de cadeia simples, o RNA dobra-se para se ligar às suas bases nitrogenadas, embora nem todas se associem.
Função do DNA e do RNA
As informações genéticas, que são as responsáveis pelas distinções entre os organismos vivos, são encontradas nas fitas de DNA.
A informação genética no DNA é o que faz com que as plantas, os animais e os humanos tenham suas formas específicas. Ela também é responsável pelas características das pessoas, como cor de cabelo, pele e tamanho.
Já o RNA é o responsável pela síntese de proteínas da célula, e cada tipo de RNA possui uma diferente função.
Tipos de RNA
Existem três tipos de RNA, o RNA mensageiro (mRNA ou RNAm), o RNA transportador (tRNA ou RNAt) e o RNA ribossômico (rRNA ou RNAr). Essas três moléculas são necessárias para a síntese proteica.
RNA mensageiro
O RNA mensageiro é uma cópia de uma das fitas do DNA, e carrega as informações genéticas do núcleo para o citoplasma de uma célula, onde será produzida a proteína.
RNA transportador
O RNA transportador está localizado no citoplasma celular e ele transporta os aminoácidos que serão utilizados na formação das proteínas até os ribossomos, onde acontecerá a síntese das proteínas.
RNA ribossômico
O RNA ribossômico está localizado no ribossomo. Quando sintetizados, os rRNA se acumulam, formando os nucléolos, que se combinam com as proteínas para originar os ribossomos.
Fonte: https://www.diferenca.com/dna-e-rna/
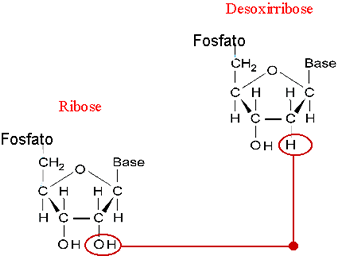


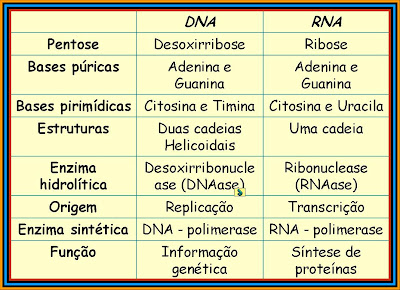
Número de cadeias: O DNA possui cadeia dupla, o RNA possui cadeia simples.
Tipo de açúcar: no DNA é a desoxirribose, no RNA é a ribose.
Bases nitrogenadas: no DNA temos adenina, timina, citosina e guanina;No RNA temos adenina, uracila, citosina e guanina.
Bases púricas são duas cadeias nitro-carbonicas (compostas prncipalmente por nitrogênio e carbono) e podem também ser nomeadas de purinas = Adenina (A) e Guanina (G)
Bases Pirimidicas são compostas por apenas uma cadeia nitro-carbonica e podem também ser nomeadas de pirimidinas = Timina (T) e Citosina (C)
3. Quais tipos de proteínas que se associam ao DNA na interfase e sua função?
A Interfase é a fase mais longa do ciclo celular. Em média, a célula passa cerca de 90% do tempo em Intérfase. Nessa fase, a célula consegue nutrientes, cresce e duplica suas moléculas de DNA. Assim, prepara-se para a divisão celular.
A interfase divide-se em três fases:
- Fase G1
- A primeira etapa da Interfase é marcada pela intensa síntese de enzimas, de RNA e no "estocamento" de proteínas compensando sua síntese descontinuada durante as etapas da Fase Mitótica. Consequentemente ocorre o crescimento celular.
- Fase S
- A segunda etapa da Intérfase é marcada, principalmente pela duplicação do material genético, logo, dobrando a ploidia da célula, fazendo que cada cromossomo apresente uma cromátide irmã.
- Além disso, na fase S, ocorre o início da duplicação dos centrossomas e o início do seus movimentos para os polos da célula.
- Fase G2
- A terceira etapa da Intérfase é marcada pela intensa síntese de proteínas e consequentemente de rápido crescimento celular, preparando a célula para a Fase Mitótica.
- O deslocamento dos centrossomas se dá por meio de dineínas e cinesinas.
- Logo que foram duplicados, os centrossomas já começam a produzir fusos astrais. Esses são associados a dineínas causando um movimento de "esteira" fazendo o centrossoma se deslocar para os polos. Ocorrem também a associação com duas cinesinas EG5 que contribuem como uma segunda força para "empurrar" os microtúbulos e deslocar os centrossomas.
- Quando o centrossoma já estiver em sua devida posição, o fuso astral relaciona-se com cinesina 14 fazendo uma força contrária à tendência de movimento, fazendo com que o centrossoma pare em determinada posição.
As fases G e S possuem estas denominações em decorrência de abreviações do inglês - G para gap (intervalo) e S para síntese.
- Fase G0
- O estágio G0 não é presente em todas as células. Nessa etapa a célula entra num estágio de repouso e não entra em processo de divisão celular.
- Essas fase é observada mais frequentemente em células musculares e nervosas.
Fase S: Duplicação DNA
ENZIMA (proteína catalizadora)
|
FUNÇÃO
|
| DNA polimerase I |
Remove os RNAs iniciadores dos fragmentos de Okazaki (atividade exonucleolítica) e preenche os espaços na fita complementar. |
| DNA polimerase III |
Catalisa o acréscimo de desoxirribonucleotídeos na forquilha de crescimento. |
| Primase(RNA polimerase) |
Catalisa a formação dos RNAs iniciadores para a síntese de DNA. |
| DNA ligase |
Catalisa a união dos fragmentos adjacentes de Okazaki. |
| Helicase |
Movimenta-se ao longo da dupla hélice de DNA utilizando a energia da hidrólise de ATP para desenrolar as duas fitas. |
| Topoisomerase I |
Corta uma fita do DNA, rotaciona essa fita sobre a outra e rejunta as extremidades, evitando o emaranhamento do DNA durante a replicação. |
| Girase (topoisomerasetipo II) |
Corta e rejunta ambas as fitas, evitando o emaranhamento do DNA durante a replicação. |

FONTE: https://djalmasantos.wordpress.com/2010/10/31/duplicacao-do-dna/
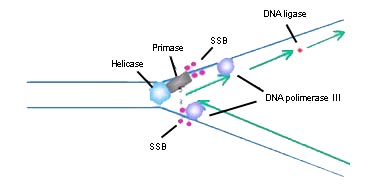
| Enzimas que actuam durante a replicação de DNA |
DNA Helicase : A função da DNA helicase é reconhecer a origem de replicação e desenrolar a dupla-hélice de DNA, na forquilha de replicação. Da sua acção resultam duas cadeias simples antiparalelas.
Single-stranded DNA binding proteins (SSB) : são proteínas que se ligam às cadeias molde separadas pela DNA Helicase, impedindo que estas se voltem a ligar, mantendo a estabilidade da forquilha de replicação. São também essenciais na reparação e recombinação de DNA.
DNA polimerase : enzima que catalisa a formação de cadeias de DNA usando as cadeias separadas como molde. Actuam no terminal 3’ da cadeia molde e só replicam na direcção 5’-3’. A DNA polimerase III é necessária para a síntese contínua da cadeia líder, enquanto que na cadeia atrasada, para além da DNA polimerase III é, também, precisa a primase para a síntese de primers iniciadores de polimerização pela DNA polimerase III, a DNA polimerase I para a remoção de primers e preenchimento de espaços e ainda a participação da DNA ligase para unir os fragmentos de Okasaki. Esta enzima pode também corrigir erros de replicação. Se foi introduzido um nucleótido errado, a DNA polimerase reconhece-o e retorna a esse ponto hidrolisando o nucleótido errado a partir da extremidade 3’. Depois de removido, a DNA polimerase prossegue o crescimento da cadeia nova.
Primase : a primase faz parte de um agregado de proteínas chamado primossoma. Esta enzima sintetiza pequenos primers que vão fornecer o terminal 3’ OH necessário para a DNA polimerase iniciar a síntese da cadeia atrasada. Este primer de RNA será mais tarde removido pela DNA polimerase I.
DNA ligase : é a enzima que une os fragmentos de Okasaki para completar a cadeia atrasada.
|
Todas as enzimas são proteínas, porém nem toda proteína é uma enzima. A enzima serve como catalisadores celulares extremamente poderosos, seu objetivo é acelerar a velocidade de uma reação química sem participar dos reagentes ou produtos; já as proteínas, são responsáveis por tudo o que o nosso corpo faz e produz, toda a proteína é funcional,ou seja, pra ser uma proteína tem que ter determinada função.
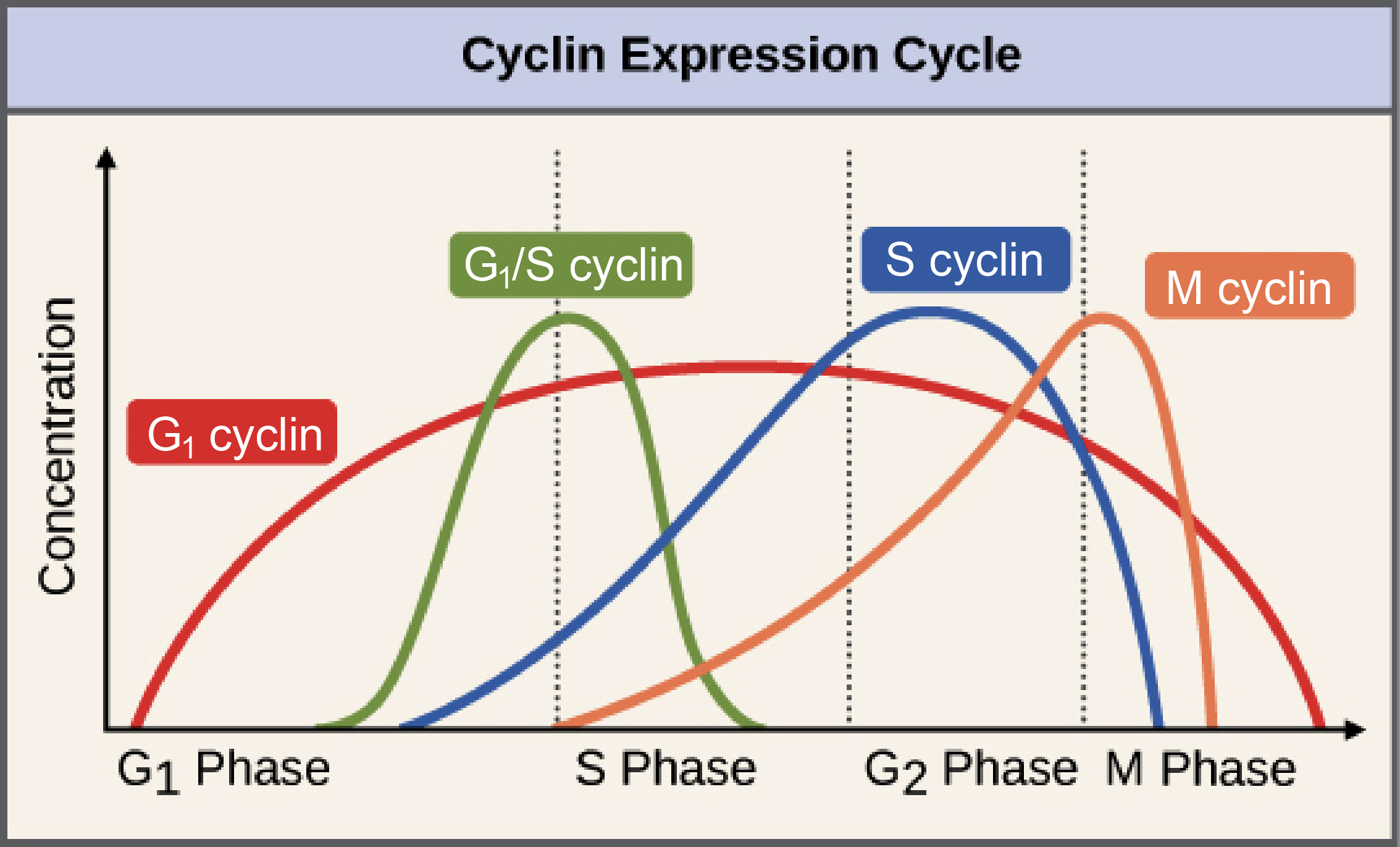
Ciclinas estão entre os mais importantes reguladores do ciclo celular. Ciclinas são um grupo de proteínas relacionadas e existem quatro tipos básicos encontrados em seres humanos e na maior parte dos outros eucariontes: ciclinas G1start subscript, 1, end subscript, ciclinas G1start subscript, 1, end subscript/S, ciclinas S, e ciclinas M.
Como os nomes sugerem, cada ciclina está associada a uma determinada fase, transição, ou conjunto de fases no ciclo celular e ajuda a conduzir os eventos dessa fase ou período. Por exemplo, a ciclina M promove os eventos da fase M, tais como a quebra do envelope nuclear e a condensação cromossômica1,2start superscript, 1, comma, 2, end superscript.
4. Explique como se dá o mecanismo de replicação do DNA durante a divisão celular.
O Processo de Duplicação de DNA. A duplicação ou replicação ocorre na intérfase da divisão celular e é dirigida pela enzima DNA polimerase. Inicia-se pela separação das fitas com a helicase. Ocorre a quebra das pontes de hidrogênio entre as bases nitrogenadas onde se juntam as sp para não retornarem e as fitas são distorcidas pela topoisomerase , formando a forquilha de replicação onde vai atuar a primase dando o start com o prime, depois vem a dna polimerase 1 na fita rápida continua e polimerase 2 na fita retardada descontinua. Na descontinua atua a ligase para colar segmentos .

http://www.uel.br/pessoal/rogerio/genetica/respostas/pratica_03.html
-
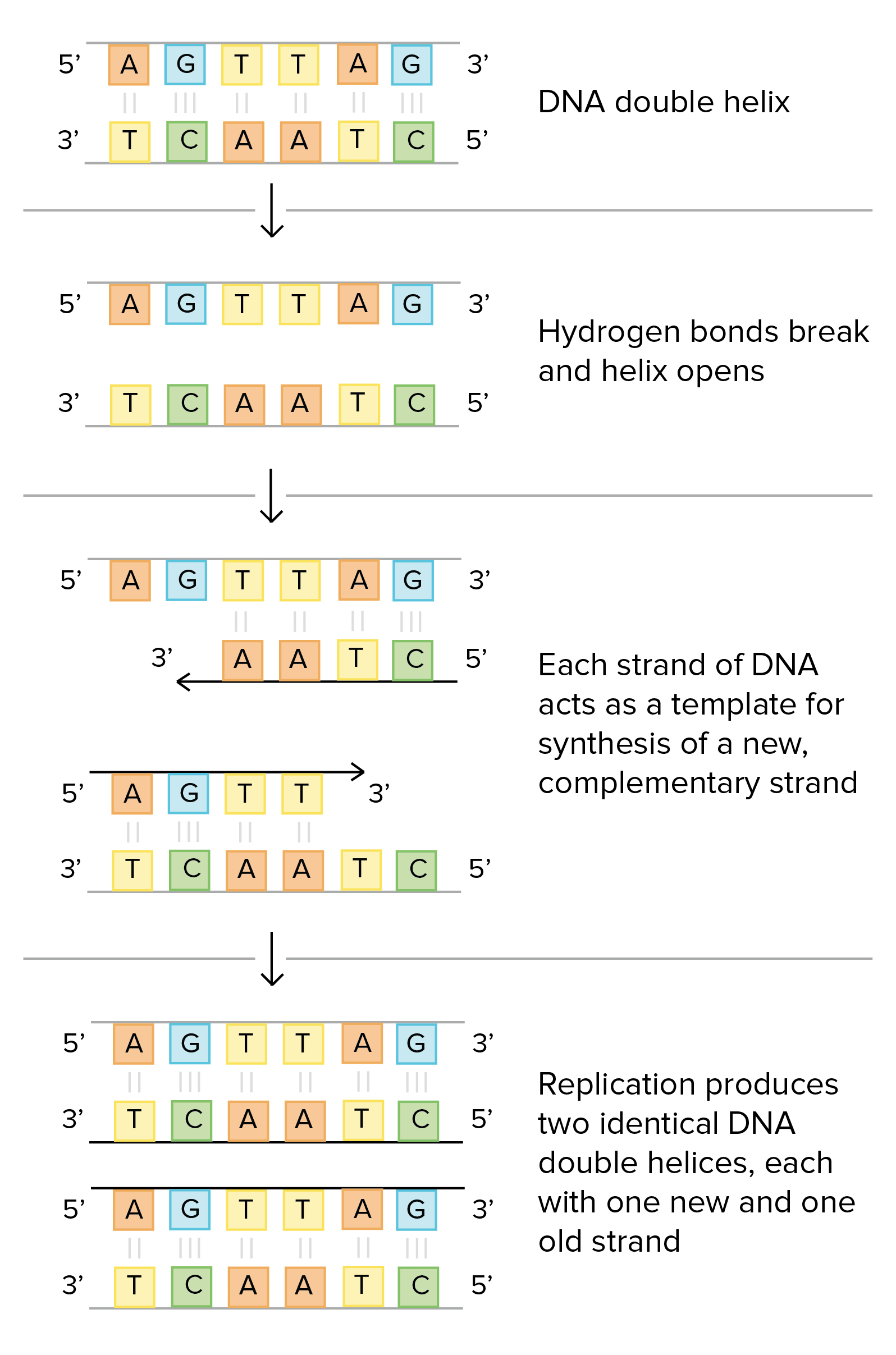
A replicação do DNA é semiconservativa. Cada fita na dupla hélice atua como modelo para a síntese de uma nova fita complementar.
-
O novo DNA é feito por enzimas denominadas DNA polimerases, que necessitam de um molde e um primer (iniciador) e sintetizam DNA na direção 5' para 3'.
-
Durante a replicação do DNA, uma nova fita (fita líder) é feita como uma peça contínua. A outra (fita tardia) é feita em pequenas partes.
-
A replicação do DNA requer outras enzimas além da DNA polimerase, incluindo DNA primase, DNA helicase, DNA ligase, e topoisomerase.
Introdução
A replicação do DNA , ou a cópia do DNA de uma célula , não é tarefa simples! Há cerca de 6,56, comma, 5 bilho˜es de pares de bases de DNA em seu genoma, os quais devem ser copiados com precisão quando qualquer uma de seus trilhões de células se divide 1start superscript, 1, end superscript .
Os mecanismos básicos da replicação do DNAsão semelhantes entre os organismos. Neste artigo, vamos nos concentrar na replicação do DNA que ocorre na bactéria E. coli, mas os mecanismos de replicação são semelhantes em humanos e outros eucariontes.
Vamos dar uma olhada nas proteínas e enzimas que realizam a replicação , vendo como elas trabalham juntas para garantir a replicação exata e completa do DNA.
O conceito básico
A replicação do DNA é semiconservativa, o que significa que cada fita na dupla hélice atua como modelo para a síntese de uma nova fita complementar.
Esse processo tem início com uma molécula e leva a formação de duas moléculas "filhas", cada uma com uma dupla hélice recém-formada contendo uma fita nova e uma velha.
De maneira resumida, essa é a replicação do DNA! Mas o que é realmente interessante é como esse processo acontece na célula.
As células precisam copiar seu DNA rapidamente e com poucos erros (para não correr riscos de ter problemas, como câncer). Para isso, utilizam uma variedade de enzimas e proteínas que trabalham juntas para garantir que a replicação do DNA seja eficiente e precisa.
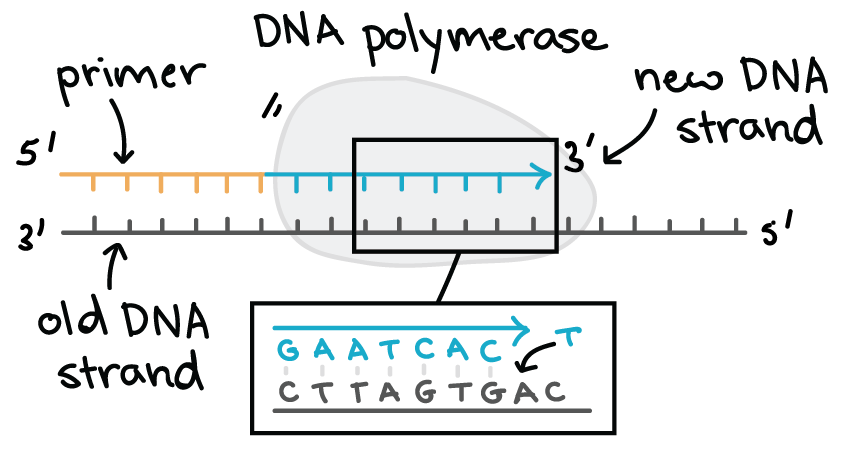
DNA polimerase
Uma das moléculas chave na replicação do DNA é a enzima DNA polimerase. DNA polimerases são responsáveis pela síntese do DNA: elas adicionam nucleotídeos, um por um, à fita crescente de DNA, incorporando somente aqueles que são complementares à fita molde.
Algumas características das DNA polimerases:
-
Sempre precisam de uma fita molde
-
Adicionam nucleotídeos somente na terminação 3' de uma fita de DNA
-
Não conseguem dar início à formação de uma cadeia de DNA; requerem uma cadeia pré-existente ou uma pequena sequência de nucleotídeos chamada de primer
-
Elas revisam (ou conferem) seu trabalho, removendo a maior parte dos nucleotídeos erroneamente adicionados à cadeia
A adição de nucleotídeos requer energia. Essa energia vem dos próprios nucleotídeos, que possuem três fosfatos ligados à sua estrutura (bastante semelhante à molécula de ATP). Quando a ligação entre os fosfatos é quebrada, a energia liberada é usada para formar uma nova ligação entre o novo nucleotídeo e a cadeia crescente.
Em procariontes como E. coli, existem duas principais DNA polimerases envolvidas com replicação do DNA: a DNA pol III (principal formadora de DNA), e a DNA pol I, que tem uma função suporte fundamental a ser estudada posteriormente.
Início da replicação de DNA
Como as DNA polimerases e outros fatores de replicação sabem onde começar? A replicação sempre começa em locais específicos no DNA, que são chamados de origens de replicação e são reconhecidos pela sua sequência.
E. coli, como a maioria das bactérias, tem uma única origem de replicação em seu cromossomo. A origem tem cerca de 2t452, t, 45 pares de bases e tem principalmente pares de bases A/T (que estão ligadas por menos pontes de hidrogênio que os pares de bases G/C), tornando as fitas de DNA mais fáceis de separar
Proteínas especializadas reconhecem a origem, ligam-se a este sítio, e abrem o DNA. Conforme o DNA se abre, duas estruturas com formato de Y, chamadas de garfos de replicação, são formadas, juntas compõem o que é chamada uma bolha de replicação. Os garfos de replicação movem-se em direções opostas à medida que a replicação acontece.
Como a replicação acontece, de fato, nos garfos? Helicase é a primeira enzima de replicação a se ligar na origem de replicação3start superscript, 3, end superscript. A função da helicase é avançar os garfos de replicação "desenrrolando" o DNA (quebrando as pontes de hidrogênio entre os pares de bases nitrogenadas).
Proteínas chamadas proteínas ligadoras de fita simples recobrem as fitas separadas de DNA próximo ao garfo de replicação, impedindo-as de ligarem-se novamente em uma hélice dupla.
Primers e primase
Polimerases de DNA somente podem adicionar nucleotídeos à extremidade 3' de uma fita existente de DNA (elas utilizam o grupo -OH livre encontrado na extremidade 3' como um "gancho", adicionando um nucleotídeo a este grupo na reação de polimerização) Como, então, a DNA polimerase adiciona o primeiro nucleotídeo em um novo garfo de replicação?
Sozinha, ela não pode! O problema é resolvido com a ajuda de uma enzima chamada primase. A primase faz um primer de RNA, ou um trecho curto de ácido nucleico complementar ao molde, que fornece uma extremidade 3' para a DNA polimerase trabalhar. Um primer típico tem cerca de cinco a dez nucleotídeos. O primer inicia a síntese de DNA, isto é, faz com que ela comece.
Uma vez que o primer de RNA está em seu lugar, a DNA polimerase o "amplia", adicionando nucleotídeos um por um para fazer uma nova fita de DNA que é complementar à fita molde.
Fita líder e fita tardia
Em E. coli, a DNA polimerase responsável pela maior parte da síntese é a DNA polimerase III. Há duas moléculas de DNA polimerase III em um garfo de replicação, cada um deles trabalhando duro em uma das duas novas fitas de DNA.
DNA polimerases podem somente fazer DNA na direção 5' para 3', e isto coloca um problema durante a replicação. Uma dupla hélice de DNA é sempre antiparalela; em outras palavras, uma fita vai na direção 5' para 3', enquanto a outra vai na direção 3' para 5'. Isto faz com seja necessário que as duas novas fitas, que também são antiparalelas a seus moldes, sejam feitas de maneiras ligeiramente diferentes.
Uma das novas fitas, a que se desloca de 5' para 3' em direção ao garfo de replicação, é a fácil. Esta fita é feita continuamente, porque a DNA polimerase está se movendo na mesma direção que o garfo de replicação. Esta fita sintetizada continuamente é chamada fita líder.
A outra nova fita, que se desloca de 5' para 3' distanciando-se do garfo, é mais complicada. Esta fita é feita em fragmentos porque, conforme o garfo avança, a DNA polimerase (que se afasta do garfo) deve separar-se e religar-se ao DNA recentemente exposto. Esta fita complicada, que é feita em fragmentos, é chamada fita tardia
Os pequenos fragmentos são chamados de fragmentos de Okazaki, em homenagem ao cientista japonês que os descobriu. A fita líder pode ser ampliada a partir de um único primer, enquanto a fita tardia precisa de um novo primer para cada um dos curtos fragmentos de Okazaki.
A equipe de manutenção e limpeza
Algumas outras proteínas e enzimas, além das principais citadas acima, são necessárias para manter a replicação do DNA funcionando adequadamente. Uma é a proteína chamada pinça deslizante, que mantém as moléculas de DNA polimerase III no lugar à medida que elas sintetizam DNA. A pinça deslizante é uma proteína em forma de anel e evita que a DNA polimerase da fita tardia escape quando ela reinicia em um novo fragmento de Okazaki4start superscript, 4, end superscript.
Topoisomerase também desempenha um importante papel na manutenção durante a replicação do DNA. Esta enzima evita que a dupla hélice de DNA à frente do garfo de replicação torne-se muito estreitamente enrolada à medida que o DNA é aberto. Ela age fazendo cortes temporários na hélice para liberar tensão, depois fecha os cortes para evitar dano permanente.
Finalmente, há um pequeno trabalho de limpeza a fazer se queremos que o DNA não contenha nenhum RNA ou lacunas. Os primers de RNA são removidos e substituídos por DNA através da atividade da DNA polimerase I, a outra polimerase envolvida na replicação. Os cortes que permanecem depois dos primers são substituídos e fechados pela enzima DNA ligase.
Resumo da replicação do DNA em E. coli
Vamos diminuir a imagem e ver como as enzimas e proteínas envolvidas na replicação trabalham juntas para sintetizar um DNA novo.
-
Helicase abre o DNA no garfo de replicação
-
Proteínas ligadoras de fita simples recobrem o DNA ao redor do garfo de replicação para evitar que o DNA se enrole.
-
Topoisomerase trabalha na região à frente do garfo de replicação para evitar enrolamento excessivo.
-
Primase sintetiza primers de RNA complementares à fita de DNA.
-
DNA polymerase III aumenta os primers adicionando nucleotídeos na extremidade 3', para fazer a maior parte do novo DNA.
-
Primers de RNA são removidos e substituídos com DNA pela DNA polimerase I
-
As lacunas entre fragmentos de DNA são fechadas pela DNA ligase.
Replicação de DNA em eucariontes
O básico da replicação de DNA é similar entre bactérias e eucariontes tais como seres humanos, mas há também algumas diferenças:
-
Eucariontes geralmente têm múltiplos cromossomos lineares, cada um com múltiplas origens de replicação. Seres humanos podem ter até 100.100, point000000origens de replicação5start superscript, 5, end superscript!
-
A maioria das enzimas de E. coli tem equivalentes na replicação eucarionte de DNA, mas uma única enzima em E. coli pode ser representada por múltiplas enzimas em eucariontes. Por exemplo, há cinco DNA polimerases humanas com funções importantes na replicação5start superscript, 5, end superscript.
-
A maioria dos cromossomos eucarióticos são lineares. Por causa da forma pela qual a fita tardia é feita, algum DNA é perdido nas extremidades dos cromossomos lineares (os telômeros) em cada evento de replicação.
5. Por que a replicação do DNA é bidirecional, explique?
Pela presença de terminal 3'OH livre, porque a medida que a forquilha abre, surgem novos iniciadores ou extremidade 3' livre na fita inferior:

"No entanto, as duas cadeias moldes são antiparalelas, em uma delas a síntese da cadeia complementar ocorre no sentido 5’ → 3’, acompanhando o sentido de abertura da forquilha. Mas, na outra cadeia, essa síntese teria de se dar em sentido inverso, ou seja, de 3’ para 5’. No entanto, as duas cadeias são sintetizadas pela polimerase III do DNA, que só catalisa o crescimento da cadeia no sentido 5’ → 3’. (Fig. 5https://edisciplinas.usp.br/pluginfile.php/2937177/mod_resource/content/2/BiologiaMolecular_texto02%20final.pdf)
"A explicação para esse aparente paradoxo é que, na forquilha de replicação, uma das cadeias é sintetizada continuamente por uma polimerase que se move no mesmo sentido do deslocamento da forquilha. Já a cadeia com polaridade inversa é sintetizada, em sentido inverso ao do deslocamento da forquilha de replicação, portanto, também no sentido 5’ → 3’. Isso só é possível porque, nesse último caso, a polimerase sintetiza curtos segmentos polinucleotídicos que crescem no sentido 5’ → 3’, que são, posteriormente, unidos para formar a nova cadeia contínua. "
A cadeia que cresce no mesmo sentido que o do deslocamento da forquilha de replicação, e cuja síntese ocorre continuamente, é chamada cadeia leading ou cadeia líder. A cadeia que cresce em sentido contrário ao do deslocamento da forquilha de replicação, e cuja síntese ocorre descontinuamente, ou seja, aos pedaços, é chamada cadeia lagging ou cadeia tardia. Esse modo de replicação do DNA, em que uma das cadeias é sintetizada continuamente e a outra, descontinuamente, é chamado de síntese semidescontínua. Costuma-se dizer também que a síntese do DNA na forquilha de replicação é assimétrica, pois em uma das cadeias (cadeia leading) ela ocorre continuamente, enquanto que na outra (cadeia lagging) ela ocorre de modo descontínuo, em fragmentos.
(idem)
6. Explique a atuação da Telomerase no processo replicativo do cromossomo.
A telomerase substitui pequenos pedaços de DNA conhecidos como telômeros , que são encurtados quando uma célula se divide por mitose .
Em circunstâncias normais, quando a telomerase está ausente, se uma célula se divide recursivamente, em algum momento a progênie atinge seu limite de Hayflick , [20] que se acredita estar entre 50-70 divisões celulares. No limite, as células se tornam senescentes e a divisão celular pára. [21] A telomerase permite que cada filho substitua o bit perdido do DNA, permitindo que a linha celular se divida sem nunca atingir o limite. Este mesmo crescimento ilimitado é uma característica do crescimento canceroso . [22]
Os telômeros protegem a extremidade do cromossomo dos danos do DNA ou da fusão com os cromossomos vizinhos. Uma transcriptase reversa (RT) é uma enzima usada para gerar DNA complementar (cDNA) de um molde de RNA , um processo denominado transcrição reversa ;
Usando TERC, TERT pode adicionar uma seqüência repetitiva de seis nucleotídeos, 5'- T TA GGG (em vertebrados, a seqüência difere em outros organismos) para a cadeia 3 'de cromossomos. Essas repetições de TTAGGG (com seus vários parceiros de ligação de proteína) são chamadas de telômeros. A região modelo do TERC é 3'-CAAUCCCAAUC-5 '. [19]
A telomerase pode ligar os primeiros poucos nucleotídeos do molde à última sequência do telômero no cromossomo, adicionar uma nova sequência de repetição do telômero (5'-GGTTAG-3 '), soltar, realinhar o novo extremo 3' do telômero ao modelo e repita o processo. A telomerase inverte o encurtamento dos telômeros .
7. Cite as enzimas relacionadas ao trabalho de duplicação do DNA e suas funções.



Helicase separa
girase desenrola
sps mão deixam as pontes voltarem
polimerase I duplica
primase constroi prime iniciador
ligase complementa e liga as falhas
8.Quais mecanismos causam mutações no DNA e sua consequência na tradução de proteínas?
Radiação
Radicais livres
Acidez pode roubar cargas das pontes de hidrogênio
Salinidade idem
Erros na copia , apesar dos muitos mecanismos de reparo
9. A molécula do DNA sofre constantes agressões, como essas agressões são reparadas?
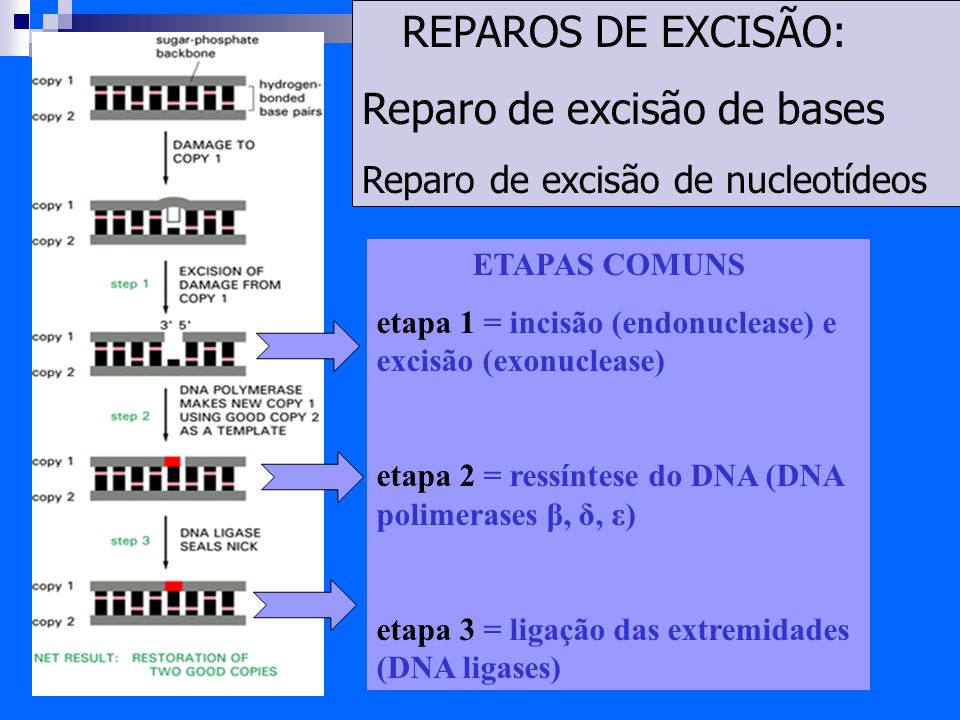


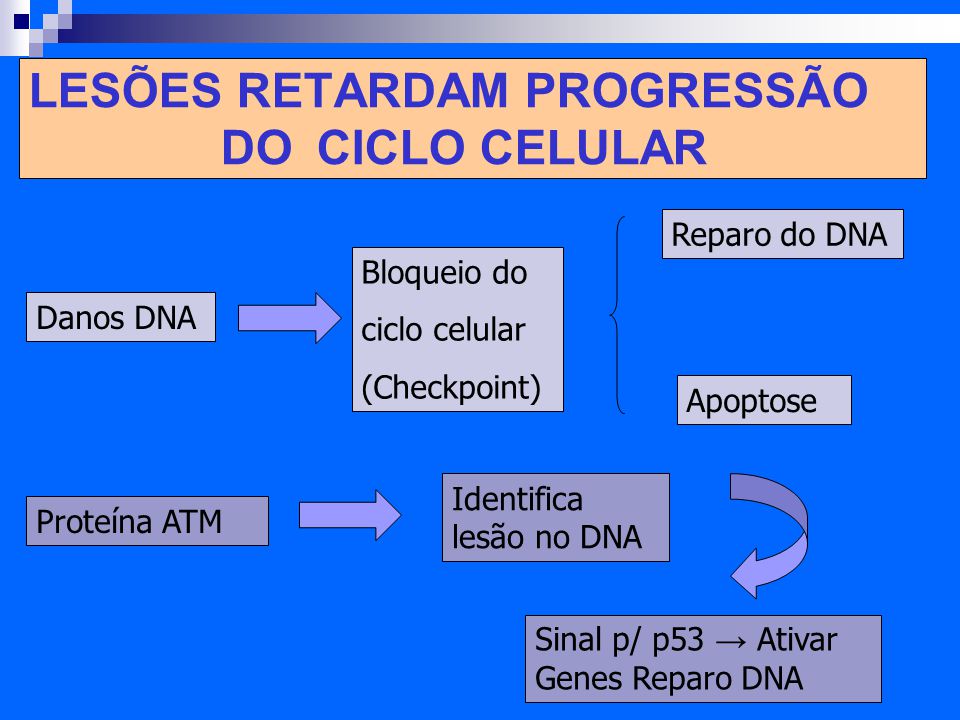 p53, p17 e outras proteinas detectam o erro
p53, p17 e outras proteinas detectam o erro
10. Quais são os tipo de RNAs gerados nas células e suas funções?
Existem três tipos de RNA, o RNA mensageiro, o RNA transportador e o RNA ribossômico.
O RNA mensageiro (RNAr) é o responsável por levar a informação do DNA do núcleo até o citoplasma, onde a proteína será produzida. Como o RNA é uma cópia fiel de uma das fitas de DNA, é a partir dessa informação que o RNA mensageiro irá determinar quais são os aminoácidos necessários para a formação de determinada proteína, pois ele possui as trincas (códons) de bases nitrogenadas que definem cada aminoácido. Por exemplo, o códon UUA determina o aminoácido leucina, o códon AUG define o aminoácido metionina e assim por diante.
O RNA transportador (RNAt) também é produzido a partir de uma fita do DNA. Esse RNA é assim chamado porque ele é o responsável por transportar os aminoácidos que serão utilizados na formação das proteínas até os ribossomos, onde haverá de fato a síntese das proteínas.
O RNA ribossômico (RNAr), chamado por alguns de RNA ribossomial, faz parte da constituição dos ribossomos. É nos ribossomos que a sequência de bases do RNA mensageiro é interpretada e a proteína, de fato, sintetizada.
11. Descreva quais as sequencias no RNA que estruturam um gene e um intron?
-
Na bactéria, os transcritos de RNA estão prontos para atuar como RNAs mensageiros e serem logo traduzidos em proteínas. Em eucariontes, as coisas são um pouco mais complexas, embora aconteça de uma forma bem interessante. A molécula diretamente feita por transcrição em uma de suas células (eucariontes) é chamada de pré-RNAm, refletindo que ela precisa passar por mais alguns passos para se tornar de fato um RNA mensageiro (RNAm). Esses são:
-
Adição de um cap 5' ao início do RNA
-
Adição de uma cauda poli-A (cauda de nucleotídeos A) ao final do RNA.
-
Separação dos íntrons ou "sequências lixo" e conexão do que sobrou , das sequências boas (os éxons).
Uma vez que esses passos estão completos, o RNA se torna um RNAm maduro. Ele pode sair do núcleo e ser usado para fazer uma proteína.A seguir achei esta analogia https://pt.khanacademy.org/science/biology/gene-expression-central-dogma/transcription-of-dna-into-rna/a/eukaryotic-pre-mrna-processing
Sua missão, caso você a aceite: decodificar a seguinte mensagem ultra-secreta. Primeiro, retire as letras "inúteis", coloridas de roxo e sublinhadas. Segundo, arranje as letras restantes em grupos de três, iniciando no começo.
THEDOGRAMAPQANANDAZAPTQMTETHEHAT
THEDOGRANANDATETHEHAT
THE DOG RAN AND A TE THE HAT
O processo pelo qual você acabou de passar é basicamente o que as suas células têm que fazer quando elas expressam um gene. Como discutimos anteriormente neste artigo, a maioria dos pré-RNAm eucariontes contêm sequências "lixo" chamadas íntrons, que são como as letras roxas na mensagem. Essas sequências devem ser removidas, e as sequências significativas (éxons), equivalentes às letras em vinho na mensagem acima, devem ser acopladas novamente para formar um RNAm maduro.
Durante a tradução, a sequência de RNAm é lida em grupos de três nucleotídeos. Cada "palavra" de três letras corresponde a um aminoácido que é adicionado a um polipeptídeo (proteína ou subunidade de proteína). Se um RNAm não tiver sofrido splicing, ele irá conter nucleotídeos a mais que não deveria, levando a uma "mensagem" de proteína incorreta. Algo similar acontece se tentamos decodificar a mensagem acima sem remover as letras roxas:
Assim como a remoção das letras roxas é essencial para se chegar à mensagem correta, o splicing também é essencial para garantir que o RNAm carregue a informação correta (e direcione a produção do polipeptídeo correto).
Cap 5' e a cauda poli-A
Ambas extremidades finais do pre-RNAm são modificadas pela adição de grupos químicos. O grupo do início (final 5') é chamado de cap, enquanto o grupo de terminação (final 3') é chamado de cauda. Tanto o cap quanto a cauda protegem o transcrito e ajudam-no a ser exportado do núcleo e a ser traduzido nos ribossomos ("máquinas" de fazer proteínas) encontrados no citosol1start superscript, 1, end superscript.
A cap 5' é adicionada ao primeiro nucleotídeo do transcrito durante a transcrição. A cap é uma guanina (G) modificada e protege o transcrito de ser quebrado. Ela também auxilia o ribossomo a se ligar ao RNAm e começar a leitura para fazer uma proteína.
Como a cauda poli-A é adicionada? O final 3' do RNA se forma de um jeito um pouco bizarro. Quando uma sequência chamada sinal de poliadenilaçãoaparece em uma molécula de RNA durante a transcrição, uma enzima corta o RNA em dois naquele ponto. Outra enzima adiciona aproximadamente 100100 -200200 nucleotídeos de adenina (A) para cotar o final, formando uma cauda poli-A. A cauda torna o transcrito mais estável e ajuda a exportá-lo do núcleo para o citosol.
Splicing do RNA
O terceiro grande evento do processamento de RNA que ocorre nas suas células é o splicing do RNA. No splicing do RNA, partes específicas do pré-RNAm, chamadas íntrons são reconhecidas e removidas por complexos proteína-e-RNA chamados de spliceossomos. Os íntrons podem ser vistos como sequências "lixo" que devem ser retiradas para que a "versão de partes boas" da molécula de RNA possa ser montada.
O que são as "partes boas"? As partes do RNA que não são cortadas são chamadas exons. Os exons são colocados juntos pelo spliceossomo para formar o RNAm maduro final, que é enviado para fora do núcleo.
Um ponto chave aqui é que são apenas os exons do gene que codificam a proteína. Não apenas os íntrons não carregam informação para formar a proteína, eles, na verdade, tem que ser removidos para que o RMAm codifique a proteína com a sequência certa. Se o spliceossomo falhar em remover um íntron, um RNAm com um "lixo" extra se formará e uma proteína errada será produzida durante a tradução.
https://www.ncbi.nlm.nih.gov/pubmed/30293940Um intron é qualquer sequência de nucleótidos dentro de um gene que é removido por processamento de RNA durante a maturação do produto de RNA final. [1] [2] Genes que codificam para histonas não possuem intron
PROCESSAMENTO DO mRNA
Comparações entre RNAs mensageiros (mRNA) extraídos do núcleo e mRNAs presentes no citoplasma revelaram que havia uma diferença muito grande no tamanho dessas moléculas e que a redução acontecia ainda no núcleo, quando partes do mRNA (os íntrons) eram removidas.
A excisão dos íntrons e reunião dos éxons para formar a fita de mRNA que será traduzida no citoplasma é um processo extremamente preciso, do corte dos íntrons e da reunião dos segmentos que correspondem aos éxons dependerá a seqüência de aminoácidos que constituirá o polipeptídio.
A redução do tamanho é apenas uma das modificações que ocorre no transcrito primário (nome que o mRNA recebe logo que é produzido) para que ele se torne uma molécula de mRNA maduro “exportável” e traduzível.
Antes de sair do núcleo, a molécula de mRNA recebe uma base modificada, denominada CAP, na extremidade 5’ (“início da fita de RNA”) e uma cauda formada por várias adeninas (centenas) na região 3’ (adição da cauda poliA no fim do mRNA). A adição desses dois elementos têm por finalidade proteger o mRNA contra degradação no citoplasma.
- D) PROCESSAMENTO ALTERNATIVO DO mRNA
Alguns genes produzem mRNAs primários que podem sofrer mais de um tipo de excisão de introns. Essas variações (denominadas de ¨splicing alternativo¨) podem ser encontradas em células de diferentes tecidos e o resultado final é que, de acordo a diferenciação, uma célula pode remover ou não um intron. Assim, um mesmo gene que se expresse em vários tecidos pode ser responsável por mais de uma cadeia polipeptídica - todas semelhantes pois se originam a partir do mesmo mRNA primário processado de modo diferente em cada tecido
Parte 2 – transcrição e tradução
12. Descreva as características que diferencial a polimerase do RNA em relação a do DNA.
A atividade das RNAs polimerases também resulta em uma cadeia de RNA que se inicia com uma extremidade 5’ e termina com um extremidade 3’, ou seja a direção de crescimento das fitas é a mesma.
Como as DNAs polimersases, as RNAs polimerases também utilizam como substrato nucleotídeos trifosfatados, de ondem provém a energia necessária para o deslocamento da enzima.
Diferente das DNAs polimerases, as RNAs polimerases não possuem atividade de revisão e correção, ou seja, não detectam erros de pareamento e não são capazes de remover nucleotídeos mal pareados. Por isso, as RNAs polimerases não necessitam de seqüências iniciadoras (primers) e não são tão precisas como as DNAs polimerases. Estima-se que as RNAs polimerases cometam um erro a cada104 nucleotídeos copiados, mas isso não traz grandes prejuízos para a célula pois o RNA não é usado como uma forma permanente de armazenamento da informação genética.
13. Como se dá o inicio do processo molecular da transcrição, explique.
A síntese do RNA começa em regiões do DNA chamadas de regiões promotoras, que são sequências específicas reconhecidas pela RNA polimerase, e direcionam a transcrição de genes.
O reconhecimento da RNA polimerase aos promotores se dá graças ao fator sigma, que liga-se à RNA polimerase fazendo com que estas tenham maior afinidade com as sequências promotoras. Os promotores contêm sequências consenso localizadas antes do início da transcrição, a distâncias específicas.
Os promotores procarióticos geralmente localizam-se na região –10 e –35 do início da transcrição e as sequências consenso mais conhecidas são o TATA box na região –10 (TATAAT) e a sequência TTGACA na região –35. Existem vários tipos de promotores e fatores sigma correspondentes e é essa variedade que permite que as funções celulares possam ser reguladas mantendo o equilíbrio das atividades celulares.
Nos eucariotos, o processo de iniciação e regulação da transcrição é muito mais complexo, envolvendo um número e diversidade maior de sequências promotoras e de fatores de transcrição (análogos ao fator sigma).
Uma importante etapa na iniciação da transcrição é a abertura da dupla fita de DNA (desenovelamento), que é feito rompendo-se as ligações entre as bases das duas fitas. É necessário que os nucleotídeos de um dos filamentos estejam disponíveis a novos pareamentos. A RNA polimerase deve desenrolar o DNA dupla hélice, formando uma bolha de transcrição, cerca de 17 pares de bases desenrolados.
14. Como ocorre o termino da transcrição?
O final da transcrição é um processo bem controlado, determinado pelo surgimento dos códons de parada ou de terminação, finalizando a síntese dessa molécula.
15. A tradução gênica é o processo no qual ocorre a síntese de uma cadeia polipeptídica. Qual o primeiro códon no mRNA que inicia o emparelhamento de um tRNA? Qual aminoácido?
UGG que é responsável pela tradução do triptofano. UGG não é um códon de parada.
A tradução gênica é o processo no qual ocorre a síntese de uma cadeia polipeptídica. Ele se inicia quando ocorre o emparelhamento de um RNAt que transporta metionina com o códon de início de tradução localizado em um RNAm que se incia com o UGG.
16. No mRNA existem três códons que simbolizam a parada da síntese, enquanto a iniciação é indicada sempre pelo mesmo códon. Quais são estes códons?
UAG.
UAA.
UGA.
17. Considere um RNA transportador cujo anticódon é CUG. O códon correspondente no RNA mensageiro e a trinca de nucleotídeos na fita do DNA que é transcrita é?
GAC no RNAm e CTG no DNA. Adenina só se liga à timina e a citosina só se liga à guanina. Também é importante lembrar que timina é encontrada apenas no DNA e uracila apenas no RNA. Sendo assim, como o anticódon do RNA transportador é CUG, no RNA mensageiro encontraremos GAC. No DNA, por sua vez, é encontrado CTG.
18. Com relação a sequencia gênica abaixo, responda?

a.Como será a molécula de mRNA transcrita ? 5' ATGCACCGA AATGAT AGAATT ACGCCC CCACAATAGA 3'
.................................................................3' UACGUGGCU UUACUA UCUAA UGCGGG GGUGUUAUCU 5'
b. Como será a sequencia da molécula de mRNA processada?
c. Qual será a sequencia de aminoácidos traduzida?
19. Considerando os diferentes códons do mRNA dado na tabela, coloque os correspondentes códons pedido:

20. O que são os operons? Quais as diferenças entre os operons dos procariotos e dos eucariotos? CONJUNTO DE GENES NOS PROCARIONTES e de alguns eucariontes, relaCIONADOS EXPRESSOS NO rnaM.
Operons são sequências lineares de DNA que as células procariontes possuem. Trata-se de um controlador do anabolismo (síntese) de enzimas pelos procariotos. Nesta postagem vamos tratar do Operon lactose (Operon lac).
O operon lac é composto basicamente por:
Ø Região Reguladora:
- uma sequência promotora.
- um gene operador.
Ø Genes estruturais:
- lacZ (gene da β-galactosidase).
- lacY (gene da permease).
- lacA (gene da transacetilase).
Em procariotos, pode haver um ou mais genes estruturais que atuam como modelos para as proteínas. Na operon lac da E. coli, existem três desses genes estruturais: lacZ, lacY e lacA. Como já mencionado, cada um desses genes é capaz de codificar uma proteína, sendo que o gene laca que codifica a transacetilase não participa do metabolismo da lactose. Os três genes estruturais do operon lac são transcritos de uma só vez em um único mRNA policistrônico, o qual é simultaneamente traduzido por vários ribossomos nas três enzimas codificadas pelo operon.
21. Em relação a tradução, o que se pode considerar da estrutura do RNA ser constituído de fita simples?



 Leia mais:
Leia mais: 















 p53, p17 e outras proteinas detectam o erro
p53, p17 e outras proteinas detectam o erro


